AIを用いたがん幹細胞の識別精度を向上させる技術を開発〜新しいがん診断法などへの応用に期待〜 東京工科大学
東京工科大学(東京都八王子市、学長:大山恭弘)応用生物学部の杉山友康教授らの研究グループ(注1)は、人工知能(AI)を用いたがん幹細胞の識別精度を大幅に向上させる技術を開発しました。
本研究成果は、2023年3月10日に学術誌「International Journal of Molecular Sciences」オンライン版に掲載されました。
本研究成果は、2023年3月10日に学術誌「International Journal of Molecular Sciences」オンライン版に掲載されました。
【ポイント】
■生成系AIを用いてがん幹細胞と非がん幹細胞を識別する技術
■別の生成系AIの出力を解析し、最良の学習用データを用いて高精度の生成系AIを作成
■予測精度が非常に悪い画像に対して、従来手法に比べ平均約5.7倍の精度向上を示した
■生成系AIを用いてがん幹細胞と非がん幹細胞を識別する技術
■別の生成系AIの出力を解析し、最良の学習用データを用いて高精度の生成系AIを作成
■予測精度が非常に悪い画像に対して、従来手法に比べ平均約5.7倍の精度向上を示した
【研究背景】
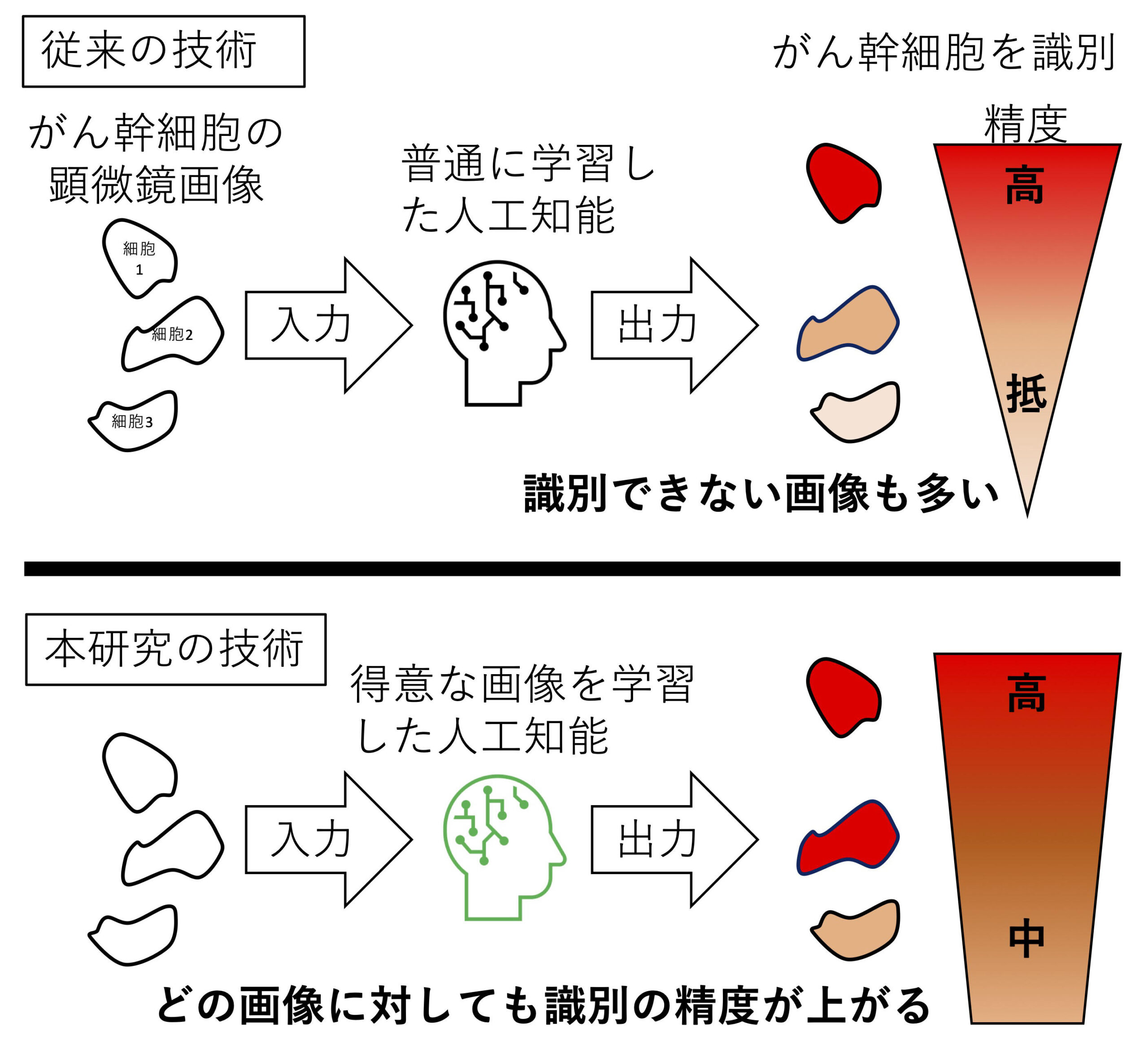
がん幹細胞はがん組織にわずかに存在し、がんの生成や亢進に重要な関連があるとされています。その実態の詳細は未解明であるものの、可塑的かつ代謝や免疫に特異な性質を有し、がん治療に対する抵抗性に影響を及ぼしていることが明らかになりつつあります。このため、がん幹細胞およびその維持を担う周辺組織を標的としたがん治療法の開発が期待されています。がん幹細胞は特徴的な発現の遺伝子やタンパク質を有し、その検出法はがん幹細胞の特定に重要な技術です。一方で、特異な細胞形態を有するとされる特徴の検出は、熟練した研究者の観察眼に負うところが大きく、技術開発は十分に進んでいません。本グループでは、生成系AIを用いてがん幹細胞を識別する技術開発に取り組み、熟練者の助け無しでがん幹細胞を識別するAIを作成しており、本研究では、その精度向上を目指した技術の開発に取り組みました。
【研究内容】
がん幹細胞1つ1つがその幹細胞の性質を維持しているかを把握するための手段として、マーカー遺伝子「Nanog」の発現を示す細胞画像を取得しました。その細胞形態の画像(画像数約7000ペア)と共に生成系AIに学習させたところ、AIは細胞形態を示す位相差顕微鏡画像の入力に対して、がん幹細胞を選別して表示しました。一方、学習に使用するデータ数や多様性に影響されず、正解の画像と比べて出力画像が9割以上正しく表示する場合と1割程度しか一致しない場合があり、AIは解析対象の画像によって得意と不得意があると考えられました。そこで、得意な画像のみを集めて生成系AIに学習させたところ、従来の生成系AIで予測した画像の精度が約6%だったところ、新技術では約40%(約5.7倍)の精度でがん幹細胞を予測することに成功しました。一方、比較的不得意な画像のみを集めて生成系AIに学習させたところ、従来手法が約90%の精度で出力するのに対して、新技術では約70%の精度となりました。(図1,2)。
【社会的・学術的なポイント】
本研究グループが開発した生成系AIは、従来の生成系AIが成しえない精度でがん幹細胞の形態を予測した点で、熟練が必要とされる高い識別能力を示したと考えられます。これらの技術をさらに発展させることで、がん診断をサポートするのに有用な手法としての応用が期待されます。また本技術で精度が向上したという事実は、AIはがん幹細胞の未特定の構造を認識した可能性を示唆しており、さらなる研究が期待されます。
【論文情報】
論文名: Deep learning of phase-contrast images of cancer stem cells using a selected dataset of high accuracy value using conditional generative adversarial networks
雑誌名:International Journal of Molecular Sciences
URL: https://www.mdpi.com/1422-0067/24/6/5323
雑誌名:International Journal of Molecular Sciences
URL: https://www.mdpi.com/1422-0067/24/6/5323
【用語解説】
(注1)東京工科大学応用生物学部4年(研究当時) 張再軍、同丸山竜人助教、同杉山友康教授、コンピュータサイエンス学部 石畑宏明教授、亀田弘之教授、岡山大学中性子医療研究センター 笠井智成准教授
■東京工科大学応用生物学部 機能性RNA工学(杉山友康・丸山竜人)研究室
ゲノムDNAやRNA中の修飾(エピジェネィック修飾)や四重鎖構造などの核酸の特殊高次構造に着目し、それらの生体機能の解析や検出方法の開発を行っています。遺伝子の働きをノックダウンする独自技術を中核として、核酸医薬の探索、遺伝子の新しい機能解明、細胞の制御、モデル動物の解析等によって新しい核酸医薬の創製を目指します。また、新属新種の細菌 Flexivirga albaを解析して、土壌環境汚染の浄化に寄与する技術開発を目指します。
[主な研究テーマ]
1.遺伝子ノックダウンした細胞の高効率評価による核酸医薬探索
2.再生モデル動物を利用した核酸医薬評価系の開発
3.がん幹細胞の画像ディープラーニングとその応用
4.六価クロム汚染浄化法の基礎研究
[研究室ウェブサイトURL]
https://sugiyama-lab.bs.teu.ac.jp
■応用生物学部WEB:
https://www.teu.ac.jp/gakubu/bionics/index.html
■応用生物学部WEB:
https://www.teu.ac.jp/gakubu/bionics/index.html
■本件の問い合わせ先
⽚柳学園 コミュニケーション企画部
担当︓⼤⽥Tel 042-637-2109
E-mail ohta(at)stf.teu.ac.jp
※(at)は@に置き換えてください